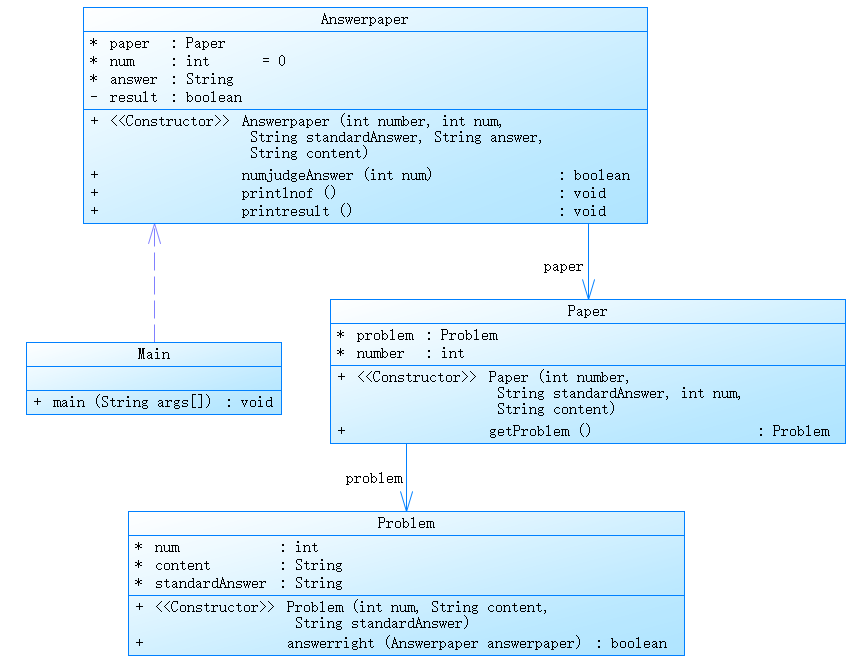
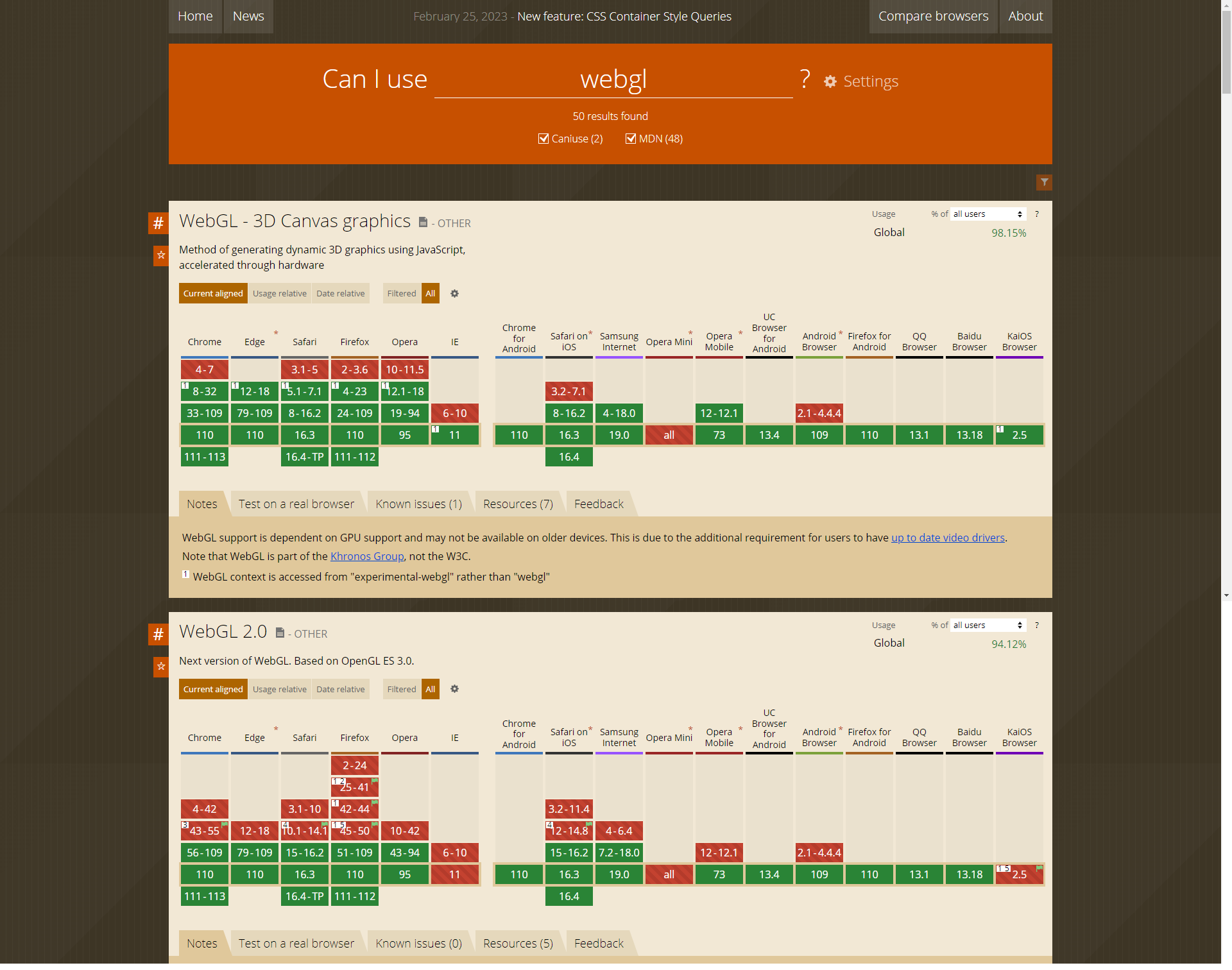
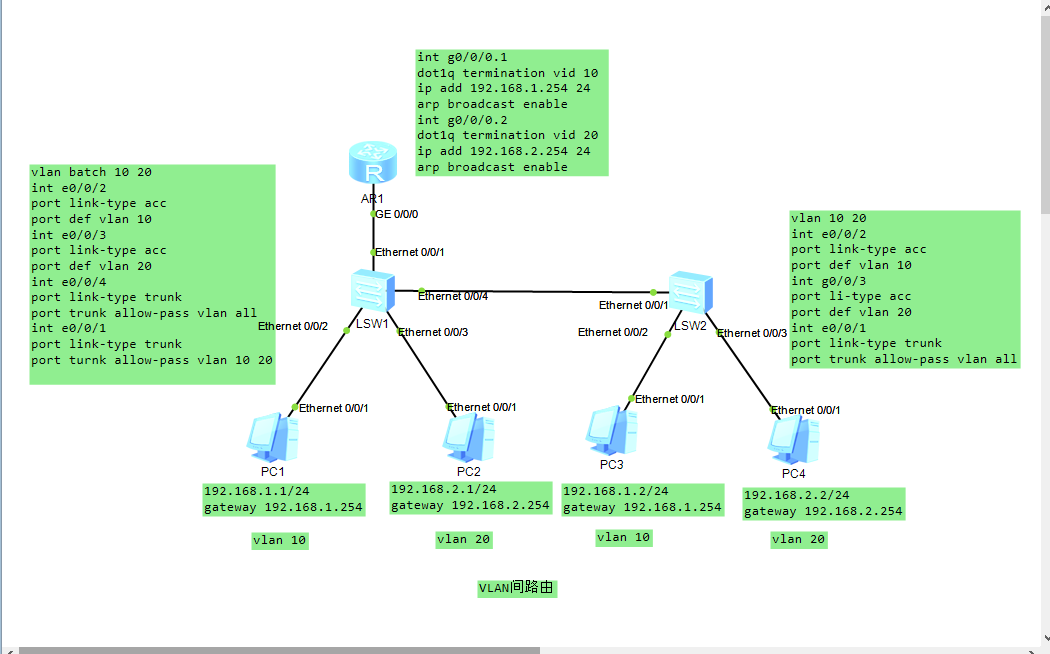
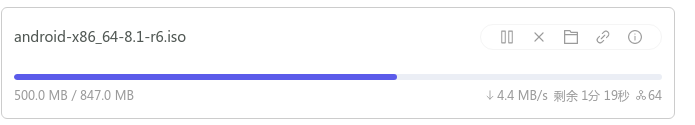
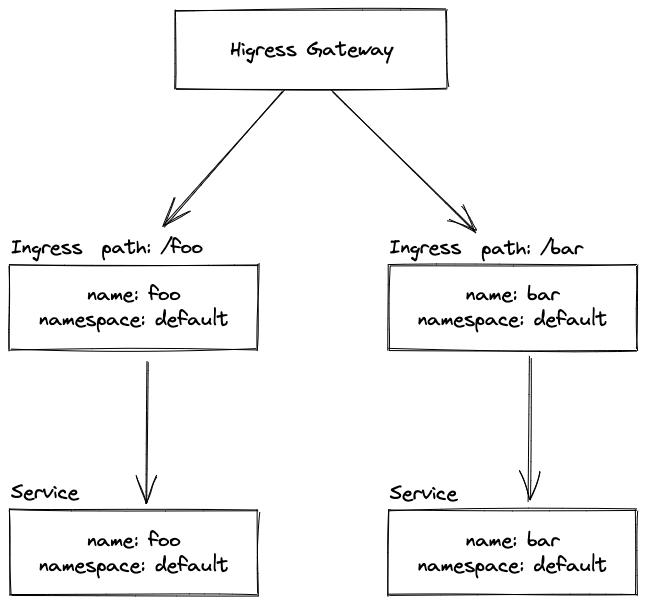
首先很容易想到的是对反串求SA和LCP,然后询问就是求起点在某个区间内的所有后缀两两LCP的最大值,可以用莫队解决,时间复杂度\(O(n\sqrt n logn)\),应该是过不了的。
考虑对原串求SAM,两个前缀的最长公共后缀(用LCS表示)其实就是它们在parent tree上对应节点的LCA的maxlen。简要证明:根据parent tree的性质,在这两个前缀对应的节点不是祖先和后代关系的情况下,这个结论是显然的;在一个是祖先一个是后代的情况下,LCS就是两个前缀中较短的那个,由于这些串都是前缀,以及后缀树的性质,较短串的长度必定等于祖先节点的maxlen。感觉白说了,因为说不太清楚但这个结论又确实很显然。
所以问题转化成了这样:有一棵\(O(n)\)个节点的树,每个节点有一个权值(maxlen)。树上有n个关键点,有q次询问,每次给出[l,r],要求只保留[l,r]中的关键点,求任意两个关键点的LCA的权值最大值。
关键点一共有\(n^2\)对,但是其实有用的只有\(O(nlogn)\)对。考虑在树上从下到上找出所有有用的关键点对。对每个点维护子树内的关键点集合,并在回溯时启发式合并。对于任意点i,假设它有两个儿子(多个是一样的),那LCA为i的关键点对只能是在左儿子的关键点集合中选一个,右儿子的集合中选一个。假设左儿子的集合是较小的,那么对左儿子集合中的任意一个点x,在右儿子集合中只有比他大的最小的点和比他小的最大的点和它配对是有用的,因为不管选哪个点配对LCA都是i,所以选跟x的编号差最小的更容易产生贡献。所以有用点对的个数和启发式合并的复杂度数量级相同,都是\(O(nlogn)\)。
找出这\(O(nlogn)\)个点之后做一个二维偏序就行了,总复杂度\(O(nlog^2n)\)。
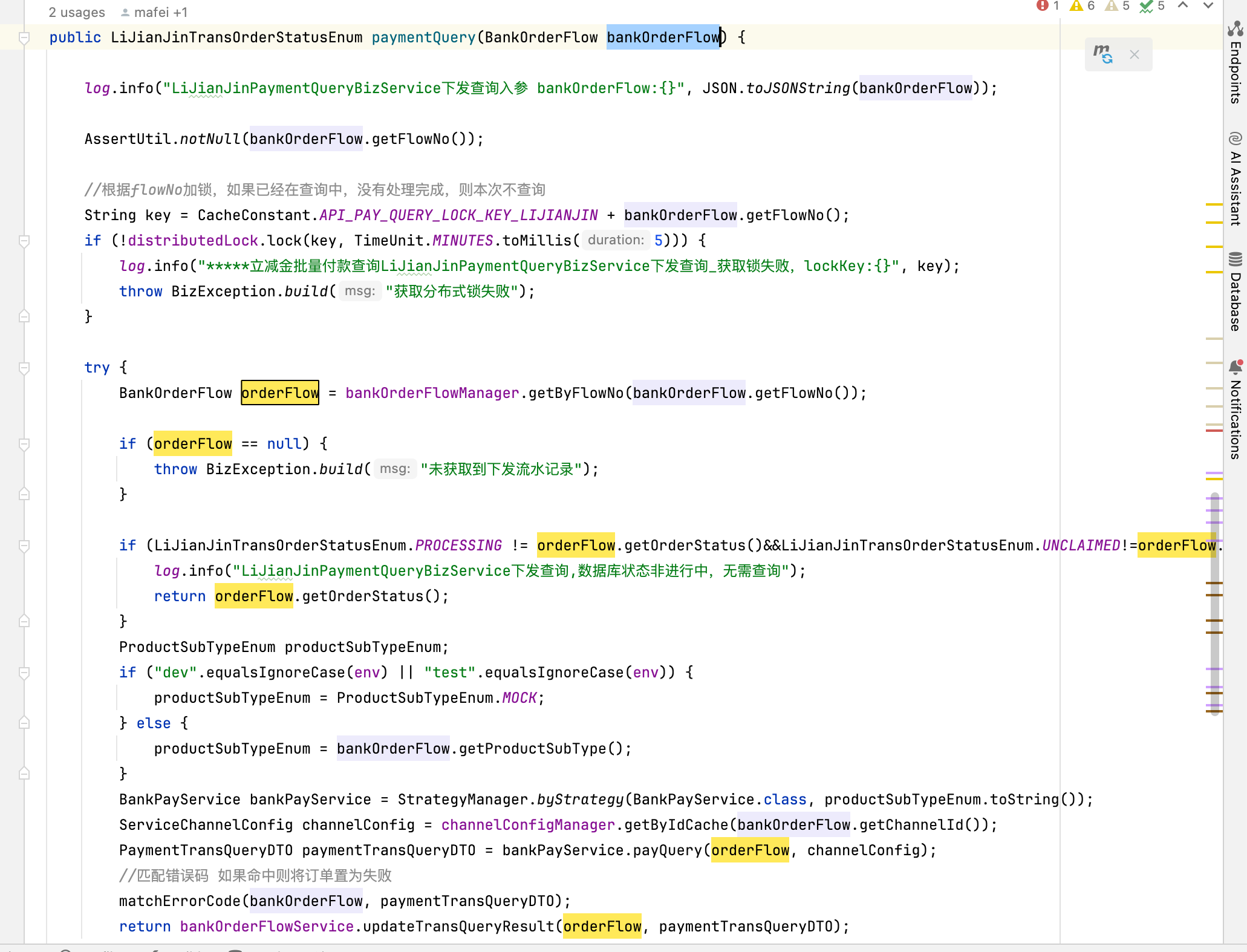
点击查看代码
#include <bits/stdc++.h>
#define rep(i,n) for(int i=0;i<n;++i)
#define repn(i,n) for(int i=1;i<=n;++i)
#define LL long long
#define pii pair <int,int>
#define fi first
#define se second
#define mpr make_pair
#define pb push_back
void fileio()
{
#ifdef LGS
freopen("in.txt","r",stdin);
freopen("out.txt","w",stdout);
#endif
}
void termin()
{
#ifdef LGS
std::cout<<"\n\nEXECUTION TERMINATED";
#endif
exit(0);
}
using namespace std;
int n,q;
string s;
char c[100010];
namespace sam
{
int lst,par[2100000],ch[2100000][30],mx[2100000],isEd[2100000],len=0;
int newNode(int val)
{
mx[++len]=val;
par[len]=isEd[len]=0;rep(i,28) ch[len][i]=0;
return len;
}
void ins(int w,int P)
{
int p=lst,np=newNode(mx[p]+1);isEd[np]=P;
while(p>0&&ch[p][w]==0) ch[p][w]=np,p=par[p];
if(p==0) par[np]=1;
else
{
int q=ch[p][w];
if(mx[q]==mx[p]+1) par[np]=q;
else
{
int nq=newNode(mx[p]+1);
rep(i,28) ch[nq][i]=ch[q][i];
par[nq]=par[q];
par[q]=nq;
par[np]=nq;
while(p>0&&ch[p][w]==q) ch[p][w]=nq,p=par[p];
}
}
lst=np;
}
}
vector <int> g[400010];
vector <pii> good[100010],que[100010];
int ans[100010];
void addGood(int x,int y,int z)
{
if(x>y) swap(x,y);
good[y].pb(mpr(x,z));
}
set <int> dfs(int pos)
{
set <int> ret;if(sam::isEd[pos]>0) ret.insert(sam::isEd[pos]);
rep(i,g[pos].size())
{
set <int> nxt=dfs(g[pos][i]);
if(nxt.size()>ret.size()) swap(nxt,ret);
for(auto it:nxt)
{
auto itt=ret.lower_bound(it);
if(itt!=ret.end()) addGood(it,*itt,sam::mx[pos]);
if(itt!=ret.begin()) addGood(it,*(--itt),sam::mx[pos]);
}
for(auto it:nxt) ret.insert(it);
}
return ret;
}
namespace st
{
int n2=1,dat[400010];
void upd(int k,int val)
{
dat[k]=max(dat[k],val);
while(k>0)
{
k=(k-1)/2;
dat[k]=max(dat[k],val);
}
}
int qry(int k,int lb,int ub,int tlb,int tub)
{
if(ub<tlb||tub<lb) return 0;
if(tlb<=lb&&ub<=tub) return dat[k];
return max(qry(k+k+1,lb,(lb+ub)/2,tlb,tub),qry(k+k+2,(lb+ub)/2+1,ub,tlb,tub));
}
}
int main()
{
fileio();
cin>>n>>q;
scanf("%s",c);s=c;
sam::lst=sam::newNode(0);
rep(i,s.size()) sam::ins(s[i]-'0',i+1);
for(int i=2;i<=sam::len;++i) g[sam::par[i]].pb(i);
dfs(1);
int x,y;
rep(i,q)
{
scanf("%d%d",&x,&y);
que[y].pb(mpr(x,i));
}
while(st::n2<n+1) st::n2*=2;
repn(i,n)
{
rep(j,good[i].size()) st::upd(good[i][j].fi+st::n2-1,good[i][j].se);
rep(j,que[i].size()) ans[que[i][j].se]=st::qry(0,0,st::n2-1,que[i][j].fi,i);
}
rep(i,q) printf("%d\n",ans[i]);
termin();
}